从牛顿在其最重要的科学著作之一《光学》中提到:“若要了解物质内部的结构情况,只要 ‘看其光谱’就可以了”,“看其光谱”一直都是人类探测物质微观结构并推演规律的重要手段。随着光谱技术在大量化学实验、分析检测、医学诊断乃至星际探索等领域中的广泛应用,海量的光谱数据不断产生,给人类“看其光谱”从而“了解结构”的传统方式带来了困难。因其依赖专家的长期经验和高精度模拟计算,传统光谱解读方法的人力与时间成本高,效率低下。发展能替代人类专家的人工智能方法,驱动机器自动解读光谱信号并反演结构信息,成为了学术界和产业界一个共同而又紧迫的“梦想”。
典型的光谱有紫外(UV)与红外(IR)吸收、拉曼(Raman)、椭圆偏振(CD)等,它们的信号来自于物质对入射光子的响应,本质上是某些量子态与光子的耦合。因此,UV与CD等光谱信号的测量是一个“信息降维”的过程,相当于物质的微观结构在某个一维频域空间的投影。不依靠专家的已有知识经验或者模拟计算中隐含的物质信息,直接利用人工智能进行“信息升维”操作,从一维的光谱信息还原得到高维的结构信息,这是一个巨大的挑战。
近期,课题组和中国科学技术大学江俊教授、加州大学Irvine分校Shaul Mukamel教授等合作,结合多尺度理论计算和机器学习技术,发展了基于二维紫外(2DUV)光谱信号智能识别蛋白质二级结构的方案,相关工作发表于《美国国家科学院院刊》(Proc. Natl. Acda. Sci. U.S.A. 2022, DOI:10.1073/pnas.2202713119)。2DUV实验利用四个超快脉冲依次轰击样品,其信号与所用脉冲的频率、偏振和入射方向及脉冲间的时间延迟相关,经由Fourier变换至频域,可将不同电子激发之间的耦合信息呈现为频率空间的二维强度分布。相比传统的一维线性光谱(如UV、CD等),2DUV具备广阔的二维特征空间,携带了远超传统一维光谱的微观结构信息。在该项工作中,作者将2DUV信号作为光谱描述符,结合卷积神经网络(CNN)和迁移学习技术,实现了基于光谱数据的二级结构识别,对同源和非同源蛋白片段的识别准确率分别达到了97%和91%,大幅度超越了基于传统一维光谱的识别效果。
为了提供高质量的蛋白质结构与对应光谱数据,如图1所示,作者基于分子动力学模拟、多组态电子结构计算、静电涨落有效哈密顿方法和Frenkel激子模型计算了14.8万具有不同二级结构的蛋白质片段结构及其LA(紫外线性吸收)、CD(紫外椭圆偏振)和2DUV光谱。
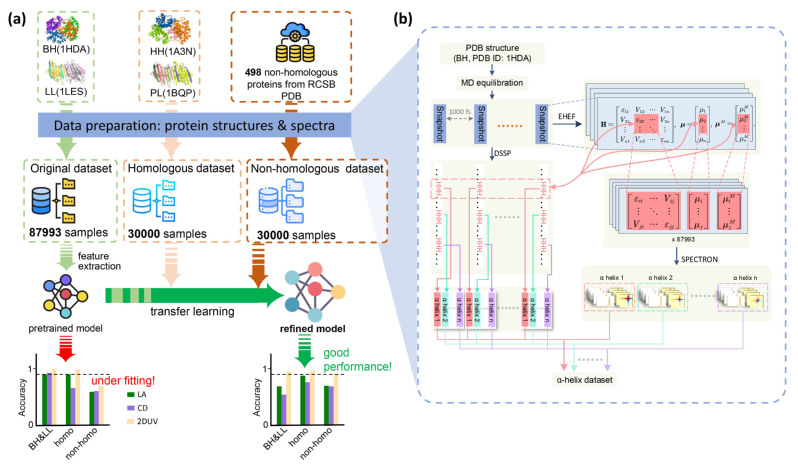
图1蛋白质片段光谱数据库构建及二级结构机器识别系统示意图。
作者将LA/CD和2DUV信号分别作为一维和二维特征描述符,训练了一维和二维卷积神经网络模型,对片段的二级结构进行识别。发现基于2DUV的二维描述符在训练难度、识别精度和数值稳定性方面均远超一维模型。梯度加权类激活映射(grad-CAM,图2)分析表明,对二级结构识别重要的光谱特征为2DUV中的非对角峰,对应于体系的激子间耦合作用,而此类作用无法利用LA和CD光谱准确表征。相对于采用LA或CD作为光谱描述符,2DUV最大的优势在于其光谱信号中显式包含蛋白质分子内位于不同酰胺键上的电子激发之间的耦合作用。相比于LA和CD谱,2DUV能够提供更高的维度容纳体系内部的相互作用特征,使得建立稳定的“结构—光谱”关联成为可能。
该项工作的意义在于,为实时、动态表征蛋白质结构做了原理论证。2018年以来,以谷歌的AlphaFold 2为代表的一系列人工智能软件,实现了从蛋白质的一维序列信息预测得到三维的结构坐标信息,打开了智能预测蛋白结构的大门。但现有的软件和算法都局限于静态情况下的蛋白质结构预测,对生命功能演化中至关重要的蛋白结构动态变化尚无成熟的办法。针对这一瓶颈难题,本次工作中算法将光谱信号视为频域序列处理,结合模式识别技术,可实现自动的光谱学信号解读。进一步结合光谱实时探测技术,人工智能算法将显著促进光谱学技术对生物大分子结构和功能演化的动态跟踪能力。当前,该结构识别模型已被部署为线上服务(http://dcaiku.com:13300),用户只需上传蛋白质片段的2DUV光谱,即可自动识别对应的二级结构。
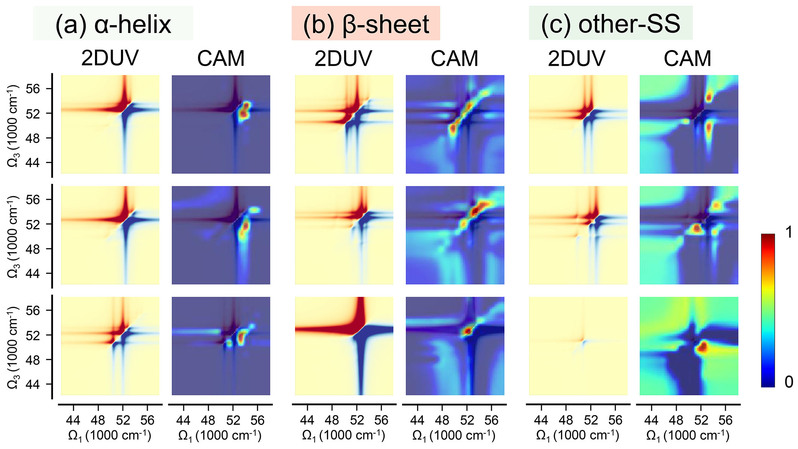
2不同二级结构的蛋白质片段的2DUV光谱(左)及对应的grad-CAM热图(右)。
该项工作得到了国家自然科学基金、科技部重点专项和合肥微尺度物质科学国家研究中心开放课题的经费支持。
